Tiempo de lectura aprox: 2 minutos, 1 segundos
Este logro es fruto de la coordinación entre la asistencia sanitaria, Salud Pública y el Área de Bioinformática Clínica de la Fundación Progreso y Salud, que interpreta las secuencias de ADN que se procesan en los hospitales
(Prensa Junta Andalucía) La Consejería de Salud y Familias ha analizado ya más de 4.200 muestras de pacientes con la enfermedad de COVID-19, con el objetivo de conocer mejor su comportamiento y poder ofrecer alternativas terapéuticas más eficaces.
En este sentido, y a través de la Dirección General de Salud Pública y Ordenación Farmacéutica, la Sanidad Pública Andaluza cuenta con un circuito estable y bien estructurado que permite la identificación y el análisis de las muestras de los pacientes, y que se enmarca en la integración de la secuenciación genómica del SARS CoV-2 en la vigilancia llevada a cabo por el Sistema de Vigilancia Epidemiológica de Andalucía. Se trata de una red colaborativa para el control epidemiológico de la pandemia que permite conocer cómo se ha transmitido el coronavirus en Andalucía desde los inicios, cuáles han sido las zonas con un mayor índice de contagio y también cómo han influido las medidas sanitarias que se han ido implantado, entre otros aspectos.
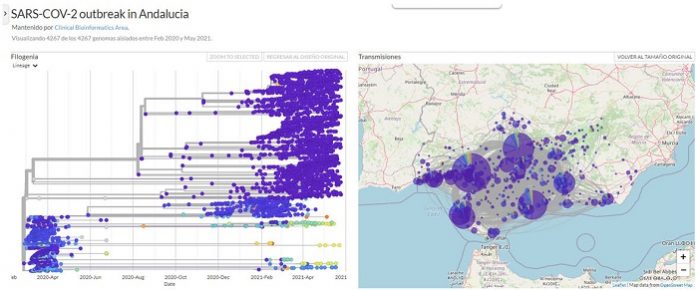
Para obtener esta información y conocer más en profundidad sobre esta enfermedad, se ha secuenciado un total de 4.267 genomas del Sars-CoV-2 en los dos hospitales de referencia designados para ello: el Hospital Virgen del Rocío de Sevilla, para secuenciar las muestras que proceden de centros hospitalarios y de Atención Primaria de la zona occidental de Andalucía (las provincias de Cádiz, Córdoba, Huelva y Sevilla); y el Hospital San Cecilio de Granada, para las que proceden de la zona oriental (Almería, Granada, Jaén y Málaga). La información obtenida ha sido analizada en el Área de Bioinformática Clínica de la Fundación Progreso y Salud, en donde se interpreta y se configura un mapa epidemiológico del virus.
Secuenciar el genoma del virus “es como leer el libro de instrucciones para descifrar su funcionamiento”, explica Joaquín Dopazo, director del Área de Bioinformática Clínica. “Esto permite comparar cada libro de instrucciones y ver semejanzas y diferencias entre ellos que se pueden relacionar con su mayor o menor transmisibilidad o incluso con diferentes sintomatologías de los pacientes”, señala.
Además, la secuenciación genómica de las muestras de pacientes permite realizar un estudio filogenético del virus, mostrando las mutaciones que experimenta a lo largo del tiempo, sus características y sus posibles relaciones con variantes de otros países o localidades, lo que facilita detectar las introducciones de virus y las cadenas de transmisión.
Un circuito estable y coordinado
Hasta llegar a los dos centros de referencia, tanto los profesionales de Microbiología como los clínicos de los hospitales y de los centros de Atención Primaria son eslabones de la cadena que trabajan de forma coordinada con el Sistema de Vigilancia Epidemiológica de Andalucía, integrado por los profesionales de Salud Pública de los distritos y hospitales, Epidemiología de las Delegaciones Territoriales Provinciales y el Servicio de Vigilancia y Salud Laboral de la Dirección General de Salud Pública y Ordenación Farmacéutica. Al final de esta cadena, el último eslabón es el Área de Bioinformática Clínica de la Fundación Progreso y Salud, donde sus profesionales analizan los datos obtenidos de la secuenciación, reportan a los centros de referencia los resultados para que estos informen al Ministerio de Sanidad de los hallazgos en la comunidad andaluza y los incorporan a las herramientas bioinformáticas para su interpretación más detallada.